| Команда | Описание |
| hisat2-build chr14.fasta chr14_index | Индексирование реферонсной последовательности. В результате было создано 8 файлов с расширением .ht2 |
| fastqc chr14.fastq | Анализ качества чтений. В результате работы получаем архив (.zip), который содержит отчет о программе в виде html файла |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr14.fastq chr14_trimmed.fastq TRAILING:20 MINLEN:50 | Очистка чтений. Обрезали с конца нуклеотды с качеством ниже 20, и убрали все последовательности длиной менее 50 нк |
| hisat2 -x chr14_index -U chr14_trimmed.fastq -S chr14_align.sam --no-spliced-alignment --no-softclip --summary-file hisat_final.txt | Триммированные чтения картированы на геном. -х – путь к индексу -U– путь к чтениям --no-softclip – запрет подрезания чтений --no-spliced-alignment – картирование без разрывов. Также результат был сохрнен в отдельный файл. |
| samtools view -b chr14_align.sam -o chr14_align.bam | Перевод из .sam формата в .bam |
| sort chr14_align.bam chr14_sorted | Сортировка выравниваний по координате в референсе |
| samtools index chr14_sorted.bam | Индексирование |
| samtools flagstat chr14_sorted.bam | Кол-во чтений, картированых на геном |
| samtools mpileup -uf chr14.fasta -go chr14_polymorf.bcf chr14_sorted.bam | Создание файла с полиморфизмом в .bcf формате |
| bcftools call -cv chr14_polymorf.bcf -o chr14_polymorf.vcf | Перевод из bcf в vcf формат |
| convert2annovar.pl -format vcf4 chr14_polymorf.vcf > chr14_polymorf.avinput | Перевод vcf-файла в формат более удобный для программы annovar |
| annotate_variation.pl -out refgene -build hg19 chr14_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация файла snp по refGene |
| -filter -out dbsnp -build hg19 -dbtype snp138 chr14_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных dbsnp |
| annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out chr14_snp chr14_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных 1000 genomes |
| annotate_variation.pl -regionanno -build hg19 -out chr14_gwas -dbtype gwasCatalog chr14_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных GWAS |
| annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 -out chr14_clinvar chr14_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных Clinvar |
Число чтений до триммирования: 8696
Число чтений после триммирования: 8562
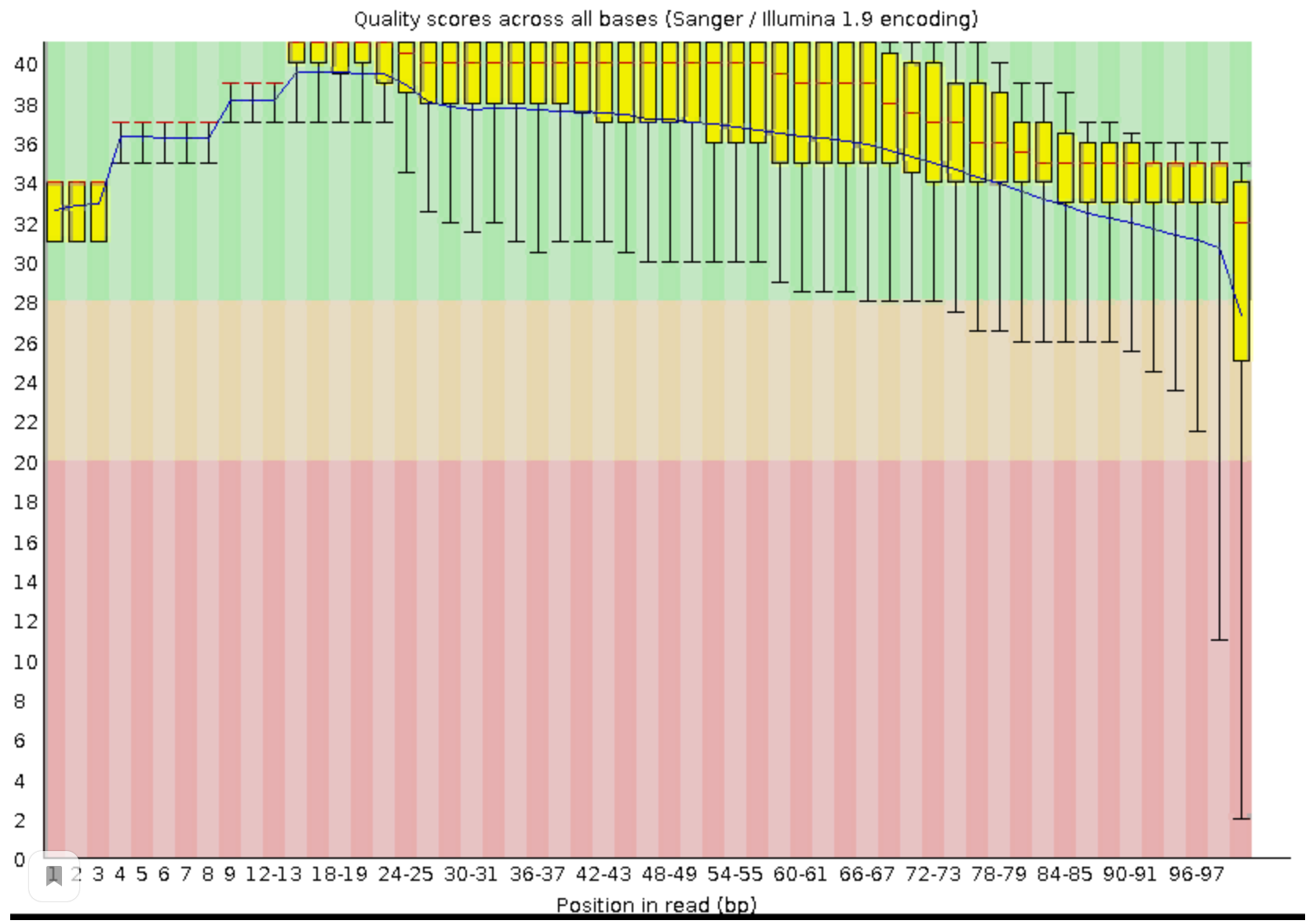
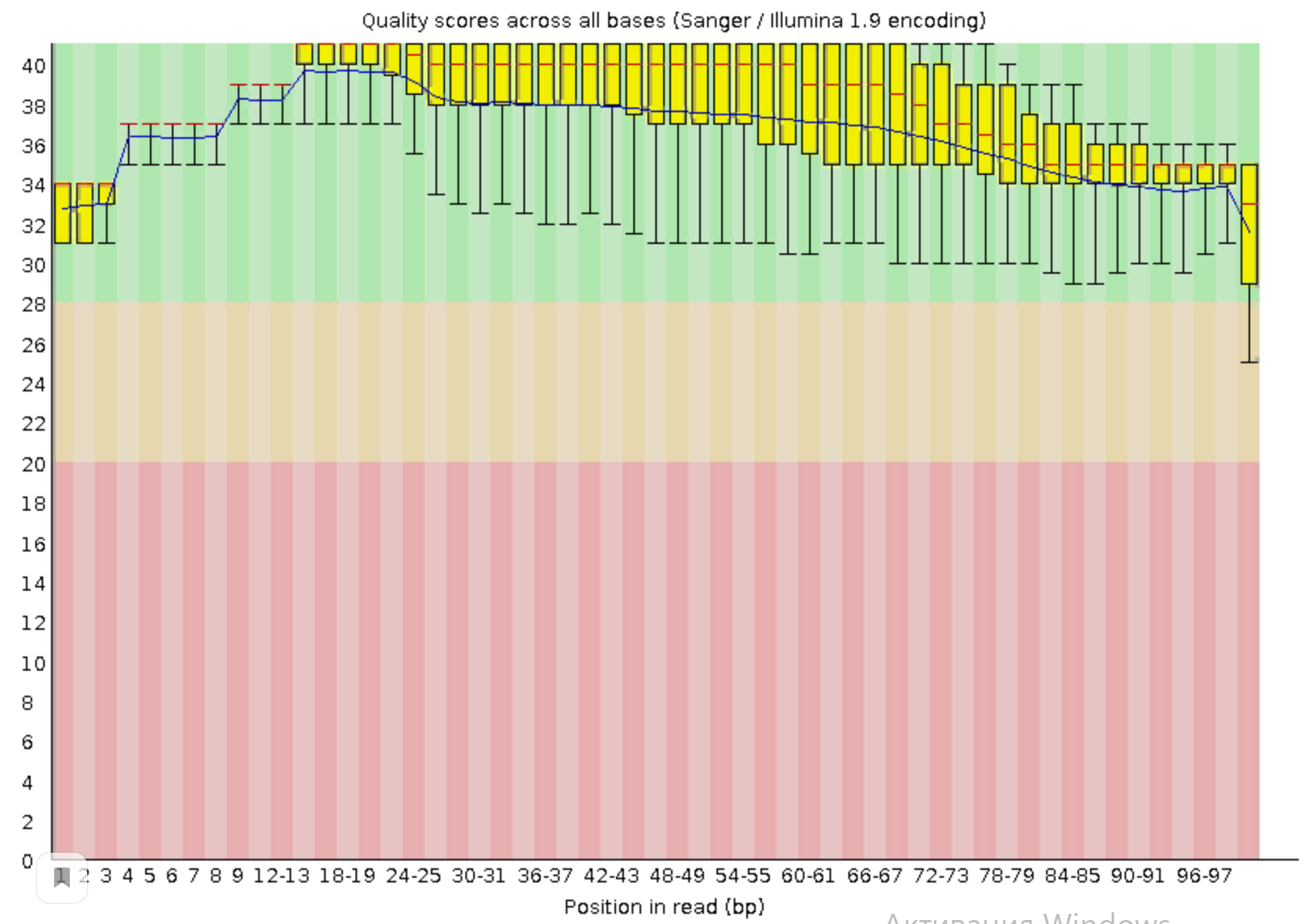
Изначальное качество чтений достаточно хорошее(больше 20). Сравнив качество чтений до и после очистки можно заметить, что ничего особо не изменилось, только в некоторых местах качество стало немного лушче. Поэтому можно сказать, что можно было бы обойтись без триммирования.

Число чтений картированных на геном= 99.75%. Ниже приведен результат работы программы.
Можно сделать вывод, что качество картирования очень высокое.
| Позиция в хромосоме | Тип полиморфизма | Референс | Чтение | Качество | Глубина |
| 81448224 | Замена | C | T | 26.0194 | 3 |
| 81448951 | Вставка | GAAAAAAAAAA | GAAAAAAAAAAAA,GAAAAAAAAAAA,GAAAAAAAAAAAAA | 84.4632 | 55 |
| 81452172 | Замена | G | A | 32.7667 | 2 |
Я получила 87 snp (из них 63 транзиций и 24 трансверсии) и 5 инделей.
RefSeq в annovar делит SNP по положению: exonic(3), splicing(1), intronic(85), ncRNA(0), UTR5(0), UTR3(1), upstream(0), downstream(0), intergenic(0).
Попали в гены PPP2R5C, RNASE9, TSHR.
| Cтрока в файле | Ген | Замена |
|---|---|---|
| 1 | RNASE9:exon4 | nonsynonymous A G |
| 51 | TSHR:exon7 | synonymous T C |
| 89 | PPP2R5C:exon16 | nonsynonymous G C |
RS имеют 83 SNP из 90 по аннотации dnsnp
0.255471 - средняя частота по выдаче 1000genomes.
Посмотрим аннотацию GWAS. (в аннотации clinvar ничего нет)
gwasCatalog Name=Prostate cancer (gene x gene interaction) chr14 21024619 21024619 A G hom 221.999 37
gwasCatalog Name=Graves' disease chr14 81451229 81451229 C T hom 221.999 71
gwasCatalog Name=Autism chr14 102360745 102360745 T C het 225.009 46
Как видно, есть три snp, имеющих клиническое значение. Первый- рак простаты (NGRG2- tumor supressor), второй- базедова болезнь, и третий- аутизм (CINP играет роль в репликации ДНК).